Software
Um die Analyse von DNA- und Chromosomen-Veränderungen zu unterstützen, entwickeln wir Algorithmen und Rechenabläufe für die Detektion mobiler DNA-Elemente aus Referenzgenomen und Hochdurchsatz-Sequenzierungen. Einige unserer Softwares zur Datenanalyse und Visualisierung sind bereits verfügbar.
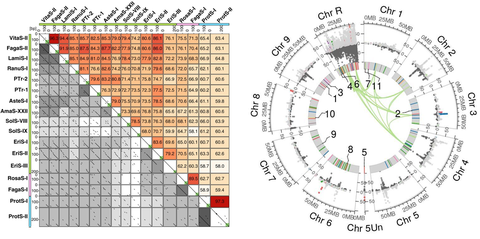
Beispielhaft wird gezeigt, wie wir Sequenzdaten analysieren und darstellen. Links: Visualisierung von Sequenzähnlichkeiten durch eine Zusammenstellung von 153 Dotplots mit unserer FlexiDot-Software. Rechts: Vergleichende Darstellung von zwei Hochdurchsatz-Sequenzierungen entlang des Referenzgenoms der Zuckerrübe durch unsere Software ECCsplorer.
ECCsplorer-Pipeline
Die in Python implementierte ECCsplorer-Pipeline (https://github.com/crimBubble/ECCsplorer) bildet die Basis für die Detektion von extrachromosomaler zirkulärer DNA (eccDNA, engl. extrachromosomal circular DNA) in beliebigen Organismen ausgehend von next-generation Sequenzierdaten. EccDNAs sind ringförmige DNAs, welche von den Chromosomen physisch getrennt und in Größen von 100 bp bis mehreren Megabasen auftreten. Sie kommen zudem in allen Eukaryonten vor und stehen oft in Zusammenhang mit repetitiven Elementen wie z.B. rDNA oder LTR-Retrotransposons. Der ECCsplorer ist eine bioinformatische Pipeline, welche automatisiert so genannte circSeq bzw. mobilome-Seq Daten (experimentelle Amplifikation zirkulärer DNA mit anschließender Illumina-Sequenzierung) analysiert und eccDNA-Kandidaten erfasst. Der ECCsplorer ist modular aufgebaut und beinhaltet sowohl einen Referenzgenom-abhängigen, als auch einen Referenzgenom-freien Ansatz zur Detektion und ermöglicht so die Nutzung für theoretisch jeden beliebigen Organsimus und für die Beantwortung einer Großzahl von unterschiedlichen biologischen Fragestellungen.
Wichtige Funktionen im Überblick:
- Modularer Ansatz für einen weiten Anwendungsbereich (Referenzgenom-abhängig, Referenzgenom-frei und komparativ)
- Nutzung von Roh-Daten möglich (Modul zur Qualitätssicherung enthalten)
- Verbessert Reproduzierbarkeit und Vergleichbarkeit von circSeq/mobilome-Seq-Studien
Die ECCsplorer-Pipeline wird in folgender Publikation beschrieben und angewandt:
Mann L., Seibt K. M., Weber B., Heitkam T. (2022): “ECCsplorer: a pipeline to detect extrachromosomal circular DNA (eccDNA) from next-generation sequencing data”, BMC Bioinformatics 23:40, doi: 10.1186/s12859-021-04545-2
SINE-finder
Das in Python implementierte SINE-Finder-Identifikationswerkzeug schuf die Grundlage für die Identifizierung von SINEs (Short Interspersed Nuclear Elements) in vielen Pflanzengenomen, darunter auch:
- Kartoffel und andere Solanaceae (in der originalen SINE-finder Publikation und hier)
- Poaceae (resultierendes Paper)
- Amaranthaceae (resultierendes Paper)
Die Identifikation von SINEs ist voraussetzend für deren Anwendung bei Inter-SINE Amplification Polymorphism (ISAP) Protokollen, wie sie durch Seibt et al. (2012) vorgestellt und durch Wenke et al. (2015) Schritt für Schritt beschrieben wurden. Unsere Partner haben derartige ISAP Protokolle ebenfalls veröffentlicht: hier und hier.
Die SINE-finder Software wird in folgender Publikation beschrieben:
Wenke T., Döbel T., Rosleff Sörensen T., Junghans H., Weisshaar B. and Schmidt T. (2011): Targeted identification of Short Interspersed Nuclear Element families shows their wide-spread existence and extreme heterogeneity in plant genomes. The Plant Cell 23(9):3117-28 read article
FlexiDot
FlexiDot logo
FlexiDot ist ein ebenenübergreifender Dotplot zur hochqualitativen Visualisierung von Einzelsequenzen, dem paarweisen Vergleich von Sequenzen, oder dem Vergleich aller Sequenzen gegen alle. FlexiDot beinhaltet Routinen für die strikte und relaxierte Handhabung von Mismatches und uneindeutigen Domänen, um die Anwendbarkeit für den Vergleich von Konsensussequenzen und anderweitig fehleranfälligen Sequenzen zu gewährleisten. Die Funtion einer benutzerdefinierten Einfärbung erleichtert die Interpretation des Dotplots und die Identifikation von wiederkehrenden Motiven, indem Informationen zur Sequenz-Annotation und zu Sequenz-Ähnlichkeiten mitabgebildet werden. Als Kollage-artiger Output ermöglicht FlexiDot die zeitgleiche Darstellung einer Vielzahl von Sequenzen und somit die Verwendung von Dotplots für Hochdurchsatzuntersuchungen.
Einige Besonderheiten im Überblick:
- hoche Flexibilität für benutzerdefinierte Anpassungen und Automatisierung
- Visualisierung von Einzelsequenzen, dem paarweisen Vergleich von Sequenzen, oder dem Vergleich aller Sequenzen gegen alle
- Hervorheben von Ähnlichkeiten durch Einfärbung
- Output als Vector- und Rastergraphiken
- Handhabung von SMRT Reads und Konsensussequenzen mit uneindeutiger Basenzuweisung (z.B. durch Alignments oder Assemblierung verursacht).
- Integration von beschreibenden Information der analysierten Sequenzen (z.B. Annotationen (gff3) oder paarweise Ähnlichkeiten)
Ein Beispiel finden Sie im Tutorial zum Hinzufügen von gff3-Annotationen zu einem Dotplot.
Siehe auch:
- Download, Beispiele und Dokumentation auf unserer github Seite
- Seibt K. M., Schmidt T. and Heitkam T. (2018): FlexiDot: Highly customizable, ambiguity-aware dotplots for visual sequence analyses. Bioinformatics doi: 10.1093/bioinformatics/bty395 read article