04.05.2023
Alternativer Treibstoff für fadenförmige Zell-Motoren
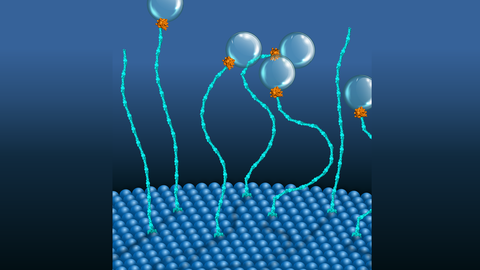
Ein molekularer Zweikomponenten-Motor, der Vesikel in die Nähe der endosomalen Membranen bringt.
Forschende aus Dresden und Bengaluru entdeckten einen einzigartigen molekularen Zwei-Komponenten-Motor, der eine Art erneuerbare chemische Energie nutzt, um Vesikel zu membrangebundenen Organellen zu ziehen.
Zellen haben die faszinierende Eigenschaft, ihr Inneres mit Hilfe winziger Proteinmaschinen, so genannter molekularer Motoren, die gerichtete Bewegungen erzeugen, zu organisieren. Die meisten von ihnen nutzen dabei einen üblichen Treibstoff, eine Art chemische Energie namens ATP. Nun haben Forschende des Max-Planck-Instituts für molekulare Zellbiologie und Genetik (MPI-CBG), des Exzellenzclusters Physik des Lebens (PoL) und des Biotechnologischen Zentrums (BIOTEC) der Technischen Universität Dresden (TUD) sowie des National Centre for Biological Sciences (NCBS) in Bengaluru, Indien, ein neuartiges molekulares System entdeckt, das eine alternative chemische Energie nutzt und einen neuartigen Mechanismus einsetzt, um mechanische Arbeit zu verrichten. Durch wiederholtes Zusammenziehen und Ausdehnen funktioniert dieser molekulare Motor ähnlich wie ein klassischer Stirlingmotor und hilft bei der Verteilung von Fracht an membrangebundene Organellen. Es ist der erste Motor, der zwei Komponenten verwendet, zwei Proteine unterschiedlicher Größe, Rab5 und EEA1, und der von GTP anstelle von ATP angetrieben wird. Die Ergebnisse wurden in der Fachzeitschrift Nature Physics veröffentlicht.
Motorproteine sind bemerkenswerte molekulare Maschinen im Inneren der Zelle. Sie wandeln chemische Energie, die in einem Molekül namens ATP gespeichert ist, in mechanische Arbeit um. Das bekannteste Beispiel ist Myosin, das unsere Muskeln bewegt. GTPasen hingegen galten bisher nicht als molekulare Krafterzeuger. Ein Beispiel ist ein molekularer Motor, der aus zwei Proteinen, EEA1 und Rab5, besteht. Im Jahr 2016 entdeckte ein interdisziplinäres Team von Zellbiolog:innen und Biophysiker:innen aus den Forschungsgruppen der MPI-CBG-Direktoren Marino Zerial und Stephan Grill und ihren Kolleg:innen, darunter der PoL- und BIOTEC-Forschungsgruppenleiter Marcus Jahnel, dass das kleine GTPase-Protein Rab5 eine Kontraktion in EEA1 auslösen kann. Diese fadenförmigen Tetherproteine können das Rab5-Protein in der Vesikelmembran erkennen und an sich binden. Die Bindung des viel kleineren Rab5 sendet eine Botschaft entlang der langgestreckten Struktur von EEA1 und macht dieses flexibel, ähnlich wie Spaghetti beim Kochen weich werden. Diese Flexibilität erzeugt eine Kraft, die das Vesikel zur Zielmembran zieht, wo es andockt und fusioniert. Ausserdem schlug das Team vor, dass EEA1 nur durch die Interaktion mit Rab5 zwischen einem starren und einem flexiblen Zustand hin- und herschalten könnte, ähnlich wie bei einer mechanischen Motorbewegung.
Hier kommt die aktuelle Studie ins Spiel, die im Rahmen der Doktorarbeit der beiden Erstautoren der Studie entstanden ist. Joan Antoni Soler aus der Forschungsgruppe von Marino Zerial am MPI-CBG und Anupam Singh aus der Gruppe von Shashi Thutupalli, ein Biophysiker am Simons Centre for the Study of Living Machines am NCBS in Bengaluru, wollten versuchen, diesen Motor experimentell in Aktion zu beobachten.
Anupam Singh verbrachte 2019 drei Monate am MPI-CBG, um die Dynamik des Proteins EEA1 zu untersuchen. „Als ich Joan kennenlernte, erklärte ich ihm die Idee, die Proteindynamik von EEA1 zu messen. Diese Experimente erforderten jedoch spezifische Modifikationen des Proteins, um dessen Flexibilität anhand seiner strukturellen Veränderungen messen zu können“, sagt Anupam. Die Expertise von Joan Antoni Soler auf dem Gebiet der Proteinbiochemie war die perfekte Ergänzung für diese anspruchsvolle Aufgabe. „Ich habe mich sehr gefreut, dass man mit der Charakterisierung des EEA1-Proteins die Frage beantworten könnte, ob EEA1 und Rab5 einen Zwei-Komponenten-Motor bilden, so wie wir zuvor vermutet hatten. Ich erkannte, dass die Schwierigkeiten, die richtigen Moleküle zu erhalten, gelöst werden könnten, indem man das EEA1-Protein so modifiziert, dass Fluorophore an bestimmte Proteinregionen binden können. Diese Modifikation würde es erleichtern, die Struktur des Proteins und die Veränderungen, die bei der Interaktion mit Rab5 auftreten können, zu charakterisieren“, erklärt Joan Antoni.
Mit den geeigneten Proteinmolekülen und der wertvollen Unterstützung von Janelle Lauer, einer leitenden Postdoktorandin in der Forschungsgruppe von Marino Zerial, konnten Joan und Anupam die Dynamik von EEA1 mit den hochentwickelten Laser-Scanning-Mikroskopen der Lichtmikroskopie-Einrichtung am MPI-CBG und am NCBS genau charakterisieren. Erstaunlicherweise entdeckten sie, dass das EEA1-Protein mehrere Male den Übergang von starr zu flexibel und wieder zurück durchlaufen kann, angetrieben allein durch die chemische Energie, die bei seiner Interaktion mit der GTPase Rab5 freigesetzt wird. Diese Experimente zeigten, dass EEA1 und Rab5 einen GTP-gesteuerten Zwei-Komponenten-Motor bilden. Um die Ergebnisse zu interpretieren, entwickelte Marcus Jahnel, einer der korrespondierenden Autoren und Forschungsgruppenleiter am PoL und BIOTEC, ein neues physikalisches Modell, das die Kopplung zwischen chemischen und mechanischen Schritten im Motorzyklus beschreibt. In Zusammenarbeit mit Stephan Grill und Shashi Thutupalli konnten die Biophysiker:innen auch die thermodynamische Effizienz des neuen Motorsystems berechnen, die mit der konventioneller ATP-getriebener Motorproteine vergleichbar ist.
„Unsere Ergebnisse zeigen, dass die Proteine EEA1 und Rab5 als molekulares Zwei-Komponenten-Motorsystem zusammenarbeiten, das chemische Energie in mechanische Arbeit umwandeln kann. Dadurch können sie eine aktive mechanische Rolle beim Membrantransport spielen. Es ist möglich, dass der Mechanismus des molekularen Motors, der die Kraft erzeugt, auch bei anderen Molekülen konserviert ist und von verschiedenen anderen zellulären Kompartimenten genutzt wird", fasst Marino Zerial die Studie zusammen. Marcus Jahnel fügt hinzu: „Ich freue mich sehr, dass wir die Idee eines EEA1-Rab5-Motors endlich testen konnten. Es ist großartig, sie durch diese neuen Experimente bestätigt zu sehen. Die meisten molekularen Motoren verwenden einen gängigen zellulären Treibstoff namens ATP. Die kleinen GTPasen verbrauchen einen anderen Treibstoff, GTP, und wurden bisher hauptsächlich als Signalmoleküle betrachtet. Dass sie auch ein molekulares System antreiben können, um Kräfte zu erzeugen und Dinge zu bewegen, wirft ein interessantes neues Licht auf diese reichlich vorhandenen Moleküle.“ Auch Stephan Grill ist begeistert: „Das ist eine neue Klasse von molekularen Motoren! Dieser Motor bewegt sich nicht wie der Kinesin-Motor, der Fracht entlang der Mikrotubuli transportiert, sondern verrichtet seine Arbeit, indem er an seinem Platz bleibt. Er ist ein bisschen wie die Tentakel eines Oktopus.“
„Das von uns verwendete Modell orientiert sich an dem des klassischen Stirlingmotors. Während der traditionelle Stirling-Motor mechanische Arbeit durch Ausdehnung und Komprimierung von Gas erzeugt, verwendet der beschriebene Zweikomponenten-Motor Proteine als Arbeitsmaterial, wobei eine veränderte Proteinflexibilität zur Krafterzeugung führt. Dieser Mechanismus eröffnet neue Möglichkeiten für die Entwicklung synthetischer Proteinmotoren“, fügt Shashi Thutupalli hinzu.
Die Autor:innen hoffen, dass diese neue interdisziplinäre Studie sowohl in der molekularen Zellbiologie als auch in der Biophysik neue Forschungsansätze ermöglichen könnte.
Originalveröffentlichung
Anupam Singh, Joan Antoni Soler, Janelle Lauer, Stephan W Grill, Marcus Jahnel, Marino Zerial, and Shashi Thutupalli: Two-component molecular motor driven by a GTPase cycle. Nature Physics (May 2023)
Link: https://doi.org/10.1038/s41567-023-02009-3
Über das MPI-CBG
Das Max-Planck-Institut für molekulare Zellbiologie und Genetik (MPI-CBG) ist eines von über 80 Instituten der Max-Planck-Gesellschaft, einer unabhängigen gemeinnützigen Organisation in Deutschland. 600 Menschen aus 50 Ländern aus den verschiedensten Disziplinen arbeiten am MPI-CBG und lassen sich von ihrem Forscherdrang antreiben, um die Frage zu klären: Wie organisieren sich Zellen zu Geweben? Die Forschung des MPI-CBG deckt dabei eine möglichst weite Spanne an verschiedenen Komplexitätsstufen ab: auf der Stufe von molekularen Netzwerken, von Zellorganellen, von Zellen, von Gewebe, Organen oder auch auf mit Blick auf ganze Organismen.
http://www.mpi-cbg.de
Über PoL
Das Exzellenzcluster Physik des Lebens (Physics of Life – PoL) der TU Dresden konzentriert sich auf die Gesetze der Physik, die der Organisation des Lebens in Molekülen, Zellen und Geweben zugrunde liegen. Am Cluster erforschen Physiker, Biologen und Informatiker gemeinsam, wie sich aktive Materie in vorgegebene Strukturen in Zellen und Geweben organisiert und damit Leben entsteht. PoL wird im Rahmen der Exzellenzstrategie durch die DFG gefördert und ist eine Kooperation zwischen Wissenschaftlern der TU Dresden und Forschungseinrichtungen des DRESDEN-concept-Verbundes, wie dem Max-Planck-Institut für molekulare Zellbiologie und Genetik (MPI-CBG), dem Max-Planck-Institut für Physik komplexer Systeme (MPI-PKS), dem Leibniz-Institut für Polymerforschung (IPF) und dem Helmholtz-Zentrum Dresden-Rossendorf (HZDR).
http://www.physics-of-life.tu-dresden.de
Über das Biotechnologisches Zentrum (BIOTEC)
Das Biotechnologische Zentrum (BIOTEC) wurde 2000 als zentrale wissenschaftliche Einrichtung der TU Dresden mit dem Ziel gegründet, modernste Forschungsansätze in der Molekular- und Zellbiologie mit den in Dresden traditionell starken Ingenieurswissenschaften zu verbinden. Seit 2016 ist das BIOTEC eines von drei Instituten der zentralen wissenschaftlichen Einrichtung Center for Molecular and Cellular Bioengineering (CMCB) der TU Dresden. Das BIOTEC nimmt eine zentrale Position in Forschung und Lehre im Forschungsschwerpunkt Molecular Bioengineering ein und verbindet zellbiologische, biophysikalische und bioinformatische Ansätze miteinander. Es trägt damit entscheidend zur Profilierung der TU Dresden im Bereich Gesundheitswissenschaften, Biomedizin und Bioengineering bei.
www.tud.de/biotec
www.tud.de/cmcb
Über das National Centre for Biological Sciences (NCBS) des Tata Institute of Fundamental Research
Der Schwerpunkt der Forschung am NCBS in Bangalore, Indien, liegt auf der Erforschung der Biologie auf allen Ebenen. Um dieses Ziel des Verständnisses lebender Systeme zu erreichen, gehen die Forschenden am NCBS biologische Fragen aus verschiedensten Richtungen an. Insbesondere das Simons Center for the Study of Living Machines innerhalb des National Centre for Biological Sciences, das Teil des Tata Institute of Fundamental Research ist, konzentriert sich auf quantitative und theoretische Ansätze zum Verstehen lebender Systeme. Das Zentrum wird daher von Wissenschaftler:innen mit unterschiedlichen Hintergründen in den Naturwissenschaften, der Mathematik und den Computerwissenschaften genutzt. Das International Centre for Theoretical Sciences (ICTS) des Tata Institute of Fundamental Research wurde 2007 gegründet und ist eine multi- und interdisziplinäre Einrichtung, die sich den theoretischen Wissenschaften widmet.
https://www.ncbs.res.in/
https://www.icts.res.in/
Zusätzliche Materialien:
Bildmaterial: https://tud.link/2nrq
Wissenschaftliche Kontakte:
Prof. Marino Zerial
Email:
Pressekontakt PoL (TUD):
Kaori Nakashima
Tel: +49 (0)351 463-41244
Email:
Pressekontakt MPI-CBG:
Katrin Boes
Tel: +49 (0) 351 210-2080
Email:
