08.11.2023
Farewell to Single Well: Vollautomatisiertes, Multiwell-smFRET ist da
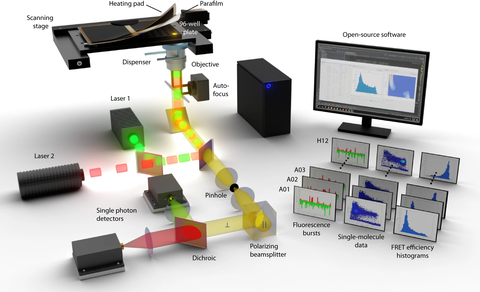
Illustration des innovativen, vollautomatisierten smFRET-Instruments für Einzelmolekül-Experimente mit hoher Durchsatzrate. Die Autoren stellen eine ausführliche Anleitung zur Erstellung oder Anpassung bestehender smFRET-Setups sowie eine umfangreiche Open-Source-Software-Suite für Datenerfassung, -verarbeitung, -analyse und -visualisierung zur Verfügung.
Die Biophysik nutzt die Physik, um unser Verständnis des Lebens auf zellulärer und molekularer Ebene voranzutreiben. Sie hat die Biologie in den letzten Jahren revolutioniert. Nun könnte die Automatisierung einer leistungsstarken Technik im Werkzeugkasten der Biophysiker:innen neue Möglichkeiten für die Biophysik eröffnen. Forschende der Schlierf-Gruppe am B CUBE - Center for Molecular Bioengineering stellten ein vollautomatisches smFRET-Instrument vor. In einer neuen Veröffentlichung im Journal Nature Communications bietet die Gruppe detaillierte Informationen zum Aufbau, eine Open-Source-Software-Suite für Messungen, Datenanalyse und Datenvisualisierung und zeigt die Möglichkeiten des neuen Systems anhand einer Vielzahl von Experimenten mit steigender Komplexität bis hin zum Screening von Medikamenten hinsichtlich Proteinfehlfaltung.
Einzelmolekül-Förster-Resonanzenergietransfer (smFRET) ist eine leistungsstarke Technik, die von Biophysiker:innen verwendet wird, um die Struktur, Dynamik und Funktion von Lebensmolekülen, hauptsächlich Proteinen, Nukleinsäuren und Lipiden, zu bestimmen. Sie wird verwendet, um festzustellen, ob zwei Positionen innerhalb einer bestimmten Entfernung voneinander liegen. smFRET hat eine Vielzahl von Anwendungen, darunter Wechselwirkungen zwischen Medikamenten und Proteinen, DNA-Reparatur, Rekombination und Replikation oder Proteinfaltung.
„smFRET-Messungen erfolgen im Nanometerbereich. Experimente sind empfindlich gegenüber äußeren Einflüssen und erfordern oft viele Bedingungen: unterschiedliche Lösungen, unterschiedliche Konzentrationen von Molekülen, ganz zu schweigen von den üblichen experimentellen Wiederholungen", erklärt Dr. Andreas Hartmann von der Schlierf-Gruppe am B CUBE. In der Realität erfordert ein typisches smFRET-Experiment oft über 50 verschiedene experimentelle Bedingungen, was Tage oder Wochen manueller Arbeit im Labor bedeutet.
„Nach über 10 Jahren Arbeit mit smFRET haben wir den Bedarf an Automatisierung erkannt. Bisher konnte kein Unternehmen ein derart automatisiertes System anbieten. Daher haben wir uns entschlossen, eines selbst zu entwickeln", sagt Prof. Michael Schlierf, Forschungsgruppenleiter am B CUBE.
Das Team legte eine Reihe von Anforderungen fest. Eine Automatisierung sollte auf jeden Fall die Präzision und Genauigkeit der Messungen nicht beeinträchtigen. Das System musste die Möglichkeit bieten, viele Bedingungen gleichzeitig zu messen. Es sollte einfach aufzubauen sein und sich gleichzeitig problemlos in bestehende Messinstrumente integrieren lassen. Schließlich musste die für den Betrieb des Instruments verwendete Software benutzerfreundlich und Open Source sein, um eine Datenanalyse ohne Expertenwissen zu ermöglichen.
Multiwell Glück
Das neue System ist vollautomatisiert und ermöglicht die Messung von bis zu 96 verschiedenen experimentellen Bedingungen, in Zukunft auch bis zu 384 Bedingungen. Das System basiert auf Multiwell-Platten, die in Laboren häufig verwendet werden. Auf diese Weise kann ein Experiment mit allen notwendigen Bedingungen und Varianten in einem Durchgang eingerichtet werden, und der Messaufbau kann unbeaufsichtigt 32 Stunden oder länger laufen, bevor die automatisch on-the-fly analysierten Ergebnisse geprüft werden.
„Unser System spart den Wissenschaftler:innen enorme Mengen an manueller Arbeitszeit. Die Experimente laufen nicht nur unbeaufsichtigt ab, sondern bieten die Möglichkeit, 50, 60 oder 90 experimentelle Bedingungen gleichzeitig einzurichten, was Wochen an Arbeit spart. Diese Zeit kann bereits für die Interpretation der Daten und die Entwicklung und Prüfung neuer Hypothesen genutzt werden", erläutert Dr. Hartmann.
Präzision in Ångströms gemessen
Das Team testete das neue System in einer gründlichen Serie von Proof-of-Concept-Experimenten, wobei jedes Experiment an Komplexität zunahm und somit immer höhere Anforderungen an das System stellte.
Zunächst verwendete das Team einen DNA-Nanomaßstab, um die Genauigkeit und Präzision des Systems zu bestimmen. „Es handelt sich um ein cleveres Werkzeug, das in den letzten Jahren als Möglichkeit zur Quantifizierung der Leistung von smFRET-Instrumenten entwickelt wurde", erklärt Dr. Hartmann. „Wir konnten zeigen, dass unser automatisiertes System hinsichtlich Präzision und Genauigkeit keine Kompromisse eingeht. Wir können innerhalb der Multiwell-Platte Abstände auf der Ångström-Ebene, also auf ein Hundertmillionstel eines Zentimeters, unterscheiden."
In einer Vielzahl von Experimenten konnte das Team zeigen, dass das System erfolgreich zur Untersuchung von DNA-Nanostrukturen, Proteinfaltung, Protein-Protein-Wechselwirkungen und Medikament-Protein-Interaktionen eingesetzt werden kann.
Für jedermann zugänglich
Diese Proof-of-Concept-Studie geht noch einen Schritt weiter und bietet gebrauchsfertige Anleitungen zur Einrichtung des Systems im Labor. „Da es sich bei smFRET um eine weit verbreitete Technik handelt, war es für uns entscheidend, allen eine Möglichkeit zur Anpassung ihrer bestehenden Setups zur Automatisierung anzubieten. Alle Komponenten des Systems sind kommerziell erhältlich und können ohne Expertenwissen installiert werden", erklärt Prof. Schlierf.
Um das System für jeden zugänglich zu machen, bietet das Team auch eine umfassende Software-Suite, die sich auf jeden Schritt des Prozesses konzentriert, von der Datenerfassung über die Verarbeitung, Analyse und schließlich die Visualisierung. Die Software ist Open Source und bietet ein umfassendes Tutorial, das es jedem ermöglicht, den Code in ihre bestehenden Arbeitsabläufe zu integrieren.
„Mit der Automatisierung vieler verschiedener Methoden in den Bereichen Biologie und Biophysik war es nur eine Frage der Zeit, bis smFRET weniger manuellen Aufwand erforderte. Wir freuen uns, dass wir dazu beitragen konnten, dies zu ermöglichen. Wir hoffen, dass dies weltweit in den Laboren viel Zeit und Ressourcen freisetzen wird. In unserem Labor hat es das auf jeden Fall getan", schließt Prof. Schlierf.
Original Veröffentlichung:
Andreas Hartmann, Koushik Sreenivasa, Mathias Schenkel, Neharika Chamachi, Philipp Schake, Georg Krainer and Michael Schlierf: An automated single-molecule FRET platform for high-content, multiwell plate screening of biomolecular conformations and dynamics. Nature Communications (October 2023)
Link: https://doi.org/10.1038/s41467-023-42232-3
Github Link: https://github.com/SchlierfLAB/autoFRET
Wissenschaftliche Ansprechpartner:
Dr. Andreas Hartmann
E-mail:
