18.07.2022
Den Zellklonen auf der Spur – bessere Diagnose mit Bioinformatik
Den Zellklonen auf der Spur – bessere Diagnose mit Bioinformatik
Den Zellklonen auf der Spur – bessere Diagnose mit Bioinformatik
Bioinformatische Methoden verbessern die Beurteilung der Klonalität bei gentherapeutisch behandelten Patient:innen
Gentherapien (GT) zielen darauf ab, die fehlende Funktionalität eines mutierten Gens auszugleichen, indem eine oder mehrere korrigierte Kopien desselben Gens in das Genom von Patient:innenzellen eingefügt werden. Für diese Einfügung werden virale Vektoren verwendet, die sich zufällig im Genom integrieren.
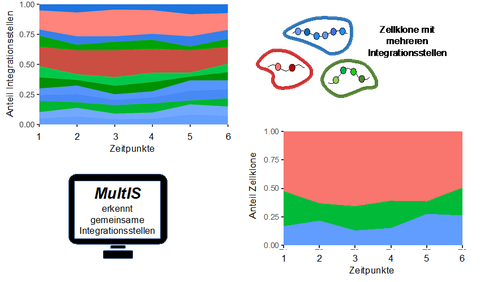
Das Tumorrisiko, welches mit diesen Integrationen verbunden ist, konnte durch Verbesserungen der Vektortechnologie weitgehend minimiert werden. Trotzdem wird die Quantifizierung der spezifischen viralen Integrationsstellen verwendet, um den relativen Anteil veränderter Zellen abzuschätzen und eventuelle Expansionsprozesse rechtzeitig zu erkennen. Allerdings kann es durch die mehrfache Integration innerhalb einer Zelle dazu kommen, dass mehr als eine Integrationsstelle pro Zellklone detektiert werden. Da im Vorfeld aber nicht erkennbar ist, ob Integrationsstellen gehäuft in einem Klon vorkommen oder unterschiedliche Klone beschreiben, kann es leicht zu einer Überschätzung der Anzahl der Klone und zu einer Unterschätzung der relativen Größe kommen.
Forschende des Instituts für Medizinische Informatik und Biometrie (IMB) der TU Dresden haben jetzt gemeinsam mit Kolleg:innen vom San Raffaele-Telethon Institut für Gentherapie in Mailand (Italien) eine bioinformatische Methode (MultIS) entwickelt, um in Zeitverlaufsdaten von Integrationsstellen typische Muster zu erkennen, die auf einen gemeinsamen klonalen Ursprung hindeuten. Mithilfe mathematischer Modelle konnte die Methode etabliert und überprüft werden. Weitere Anwendungsbeispiele für in vitro und in vivo Daten belegen die breite Einsetzbarkeit und bestätigen außerdem, dass klonale Assoziationen von Integrationsstellen innerhalb eines Zellklones auch im Zeitverlauf einer gentherapeutischen Maßnahme identifiziert werden können. Damit wird es perspektivisch möglich sein, frühzeitig die klonale Dynamik bei Patient:innen nach Gentherapie einzuschätzen und gegebenenfalls angemessene Gegenmaßnahmen einzuleiten. Unsere Ergebnisse zeigen, dass es die vorgestellte Methodik ermöglicht, systematische Verzerrungen bei der klonalen Quantifizierung zu überwinden und damit eine wesentliche Voraussetzung für die bessere Bewertung der Sicherheit und langfristigen Wirksamkeit von Gentherapien darstellt.
Die Ergebnisse dieser Arbeit wurden am 28. Juni 2022 in Nature Communications veröffentlicht (S. Wagner et al., Clonal reconstruction from co-occurrence of vector integration sites accurately quantifies expanding clones in vivo, https://www.nature.com/articles/s41467-022-31292-6).
Weitere Informationen:
PD. Dr. Ingmar Glauche
Institut für Medizinische Informatik und Biometrie
TU Dresden, Medizinische Fakultät Carl Gustav Carus
Fetscherstr. 74, 01307 Dresden
Partner:innen:
Daniela Cesena, Andrea Calabria, Eugenio Montini
https://research.hsr.it/en/institutes/san-raffaele-telethon-institute-for-gene-therapy/safety-of-gene-therapy-and-insertional-mutagenesis/eugenio-montini.html
Twitter:
https://twitter.com/NatureComms/status/1544591478315515904?t=gayVe8kdcZIk0oM5Hcu8Mg&s=19
