Oct 14, 2020
Tumor characterization using mass spectrometry - new community supports research projects in Dresden
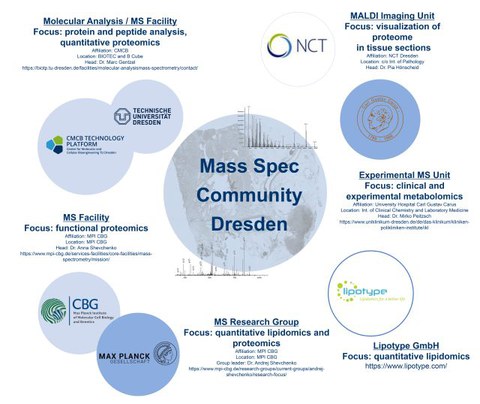
Mass spectrometry is a versatile and powerful analytical method that is becoming increasingly important for tumor characterization in addition to common DNA and RNA analyses. For example, it can be used to determine the exact protein or lipid composition of tumor tissue in order to better differentiate between different tumor types, to discover biomarkers or to make new tumor classifications. To make the highly complex mass spectrometric analysis possibilities more accessible for research, scientists from various Dresden institutions - Max Planck Institute of Molecular Cell Biology and Genetics, Center for Molecular and Cellular Bioengineering (CMCB, TU Dresden), Lipotype GmbH, National Center for Tumor Diseases Dresden (NCT/UCC), University Hospital Carl Gustav Carus Dresden - have joined forces to form the "MS Community Dresden".
Together, the community has more than 20 different mass spectrometry instruments and a wide range of specialized expertise in the field of research and diagnostics. The aim of the grouping is - in addition to independent joint research activities - to facilitate access to comprehensive mass spectrometric analyses for scientists of the participating institutions and to support them in planning projects.
"We would like to support scientists who are planning a project or study in this field as consultants and pilots. The members of the community have a wealth of experience in different approaches to mass spectrometry and the fields of proteomics, metabolomics, lipidomics and MALDI imaging. They are therefore able to coordinate workflows and smoothly integrate analyses at equipment and in units of different institutions and propose analytical methods," says Dr. Pia Hönscheid, member of the MS community and scientist at NCT/UCC Dresden. Especially for issues that require two or more approaches, such as linking with transcriptomics or genomics, the so-called MultiOmics projects, a common approach is essential. Researchers can submit their requests both through existing service facilities of the participating institutions and through joint research collaborations.
Anyone interested in receiving support from the MS Community can contact MS Community members directly or present their research project, for example at the biweekly (online versions are also possible) Journal Club. We also welcome all those who want to learn about new developments in the field of mass spectrometric research.
Mass spectrometry covers a wide range of applications and is particularly well established in the fields of lipidomics, metabolomics, proteomics and polymer research. Biomolecules are identified and quantified by their exact mass and by the exact mass of their fragments. The field of application ranges from small molecules such as lipids, sugars and metabolites to megadalton-sized protein complexes. The analysis can be "targeted" to specific molecules or "non-targeted" to the global analysis of the metabolome/lipidome or proteome including the discovery and identification of new molecules. Methods of FFPE-based peptide and protein analysis are also established for clinical application and can be applied to translational projects, e.g. in practice-oriented cancer research.
Contact:
Dr. Marc Gentzel
CMCB Facility Leader Mass Spectrometry
Tel: 0351-463 40212/-210
Email: marc.gentzel@tu-dresden.de