Korrelation zwischen der Centrosomengröße und dem Aufbau der mitotischen Spindel
In den meisten tierischen Zellen wird die mitotische Teilung durch ein bipolar angeordnetes Paar Centrosomen durchgeführt. Centrosomen sind hauptsächlich verantwortlich für die Ausbildung von Mikrotubuli, welche die Teilungsspindel am Zellkortex verankern (astrale Mikrotubuli) und am genetischen Material binden können um es zu trennen (Kinetochormikrotubuli). Wie vorherige Studien gezeigt haben, kann das Volumen der Centrosomen im C. elegans Embryo sehr variabel sein und sich z.B. an die Größe des Embryos anpassen [1,2]. Obwohl lichtmikroskopische Analysen einen Zusammenhang zwischen Centrosomengröße und der Anzahl ausgebildeter Mikrotubuli vermuten lassen, fehlen hochauflösende Ergebnisse auf ultrastruktureller Ebene um eine quantitative Skalierung zu beschreiben.
Unser Ziel ist es herauszufinden wie die Größe der Centrosomen den ultrastrukturellen Aufbau der mitotischen Spindel beeinflusst und in wie fern diese Korrelation sich auf die Anzahl und Verteilung der nukleierten Mikrotubuli auswirkt.
Dafür benutzen wir einen korrelativen licht- und elektronenmikroskopischen Ansatz (CLEM) in dem die Teilungsdynamik an lebenden Embryonen verfolgt wird. Anschließend wird die Struktur der Proben zu einem spezifischen Zeitpunkt durch Hochdruckgefrieren in flüssigen Stickstoff konserviert und es können durch Elektronentomographie 3D Modelle der Teilungsspindel rekonstruiert werden [3].
Zur Zeit konzentrieren wir uns auf die Untersuchung des Proteins SAS-4, welches für die Verdopplung der Centriolen mitverantwortlich ist. Ein Abbau des Proteins führt zu stark asymmetrischen Spindeln in der zweiten embryonalen Teilung mit Centrosomen unterschiedlicher Größe [4]. Wir zeigen die erste 3D Rekonstruktion einer asymmetrischen Mitosepindel im Zweizellstadium. Im Vergleich zum Wildtyp führt hier das Ausschalten von SAS-4 zu einer Reduzierung der Centrosomengröße um circa 70 % und einer damit verbundenen Verringerung der ausgebildeten Mikrotubuli um mehr als 90 %.
[1] Decker et al. 2001. Curr. Biol. 21, 1259-1267.
[2] Greenan et al. 2010. Curr. Biol.. 20, 353-358.
[3] Müller-Reichert et al. 2003. J. Microsc. 212:71-80.
[4] Kirkham et al. 2003. Cell. 112, 575-587.

Figure 1: Live-cell imaging of sas-4 (RNAi) C. elegans two-cell embryos. Sum projection of confocal stacks using SPD-2::GFP to visualize centrosomes (green) and H2B::mCherry to mark chromosomes (red). The anterior side of embryos is orientated to the left. Scale bar = 10 µm. A: Wild type embryos exhibit equally sized centrosomes and perfectly manage mitotic divisions. B: Full depletion of sas-4 by RNAi injection (24h) causes monopolar spindles in two-cell embryos uncapable of proper chromosome segregation in subsequent mitotic divisions. C: Partial depletion of sas-4 by RNAi feeding (24h) leads to an intermediate phenotype showing highly asymmetric spindles within the P1 cell resulting in a small (arrow) and a normal sized centrosome.
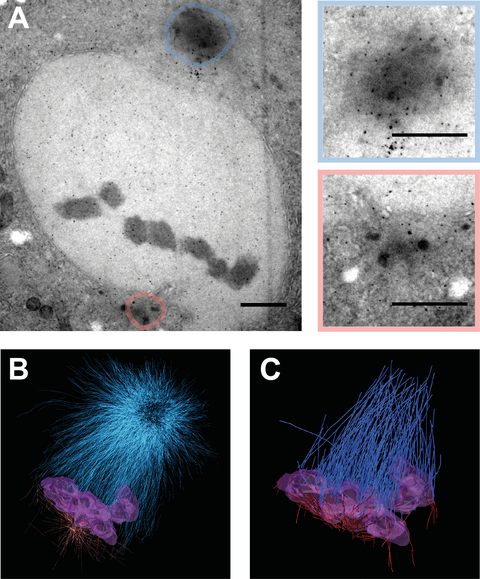
Figure 2: Electron Tomography of sas-4 (RNAi) two-cell embryos. TEM images of high pressure frozen and sectioned C. elegans two-cell embryos at metaphase obtaining an asymmetric spindle in P1 cell. A: Asymmetrical P1 spindle at metaphase revealing a large centrosome in blue and a small centrosome in red. Color coded magnifications of centrosomes depicted on the right. Scale bar = 1 µm. B: 3D reconstructed volume of spindle shown in A. Blue: Microtubules of large pole, Red: Microtubules of small pole, Violet: chromosomes C: Filtered kinetochore microtubules from 3D reconstruction shown in B.