Mar 07, 2024
Konzeption eines generischen Datenharmonisierungsprozesses für OMOP Common Data Model
Die Nutzung von sogenannten Real-World Daten für die Forschung wird immer wichtiger, um Einblicke in die reale Versorgung von Patient:innen im Gesundheitssystem zu erlangen und auf dieser Grundlage neue Erkenntnisse für die Diagnose, Behandlung und Prävention von Krankheiten zu gewinnen. Um einen umfassenden Überblick über die Krankheitshistorie von Patient:innen zu erhalten, ist die Verknüpfung klinischer Daten in Form von Daten der Datenintegrationszentren (DIZ) der Medizininformatik-Initiative (MII) mit ambulanten Daten in Form von Routinedaten der gesetzlichen Krankenversicherung (GKV) sinnvoll. Zur Sicherstellung der semantischen und syntaktischen Interoperabilität beider Datensätze ist eine Überführung in ein standardisiertes Datenmodell, wie beispielsweise in das von der Observational Health Data Sciences and Informatics (OHDSI) Community bereitgestellte Observational Medical Outcomes Partnership (OMOP) Common Data Model (CDM) erforderlich. Leider existiert jedoch kein detaillierter Leitfaden, der es Forscher:innen ermöglichen würde, einen einheitlichen Prozess zur Datenharmonisierung in OMOP CDM zu durchlaufen. Das Ziel unserer Arbeit war es daher, einen generischen Datenharmonisierungsprozess für OMOP CDM zu konzipieren. In diesem Kontext konzentrierten wir uns auf die folgenden drei Forschungsfragen:
- Welche Prozessschritte müssen bei der Harmonisierung klinischer Daten oder GKV-Routinedaten in OMOP CDM durchgeführt werden?
- Welche OHDSI-Tools wurden von anderen Forscher:innen zur Unterstützung der Harmonisierung klinischer Daten und GKV-Routinedaten in OMOP CDM verwendet?
- Welche Reihenfolge der identifizierten Prozessschritte gilt es zu beachten?
Um einen Überblick über den aktuellen Stand des methodischen Vorgehens bei der Datenharmonisierung klinischer Daten und GKV-Routinedaten in OMOP CDM zu erhalten, wurde eine Literaturrecherche durchgeführt. Im Rahmen der Datenextraktion fand für jede eingeschlossene Publikation die Extraktion verwendeter Prozessschritte und deren chronologischer Reihenfolge sowie verwendeter OHDSI-Tools statt. Anschließend erfolgte ein Vergleich der Ergebnisse, um eine generische Abfolge der Prozessschritte abzuleiten.
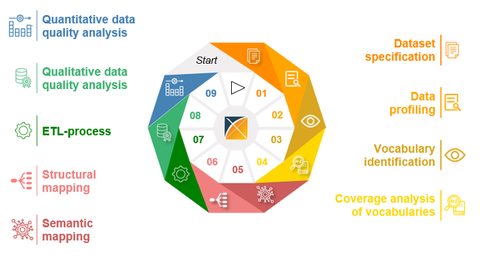
Aus insgesamt 23 eingeschlossenen Publikationen ließ sich ein generischer Datenharmonisierungsprozess konzipieren, welcher sich aus neun Prozessschritten in der folgenden Reihenfolge zusammensetzt:
- Datensatzspezifikation
- Datenprofilierung
- Vokabularidentifikation
- Abdeckungsgradanalyse Vokabulare
- Semantisches Mapping
- Strukturelles Mapping
- ETL-Prozess
- Qualitative Datenqualitätsanalyse
- Quantitative Datenqualitätsanalyse
Des Weiteren konnten sieben OHDSI-Tools identifiziert werden, welche die Durchführung von fünf Prozessschritten unterstützten.
Unser konzipierter generischer Datenharmonisierungsprozess für OMOP CDM kann als Leitfaden verwendet werden, um andere Forscher:innen bei der Harmonisierung von Quelldaten in OMOP CDM zu unterstützen. Als zukünftige Arbeit wird die Anwendbarkeit des generischen Datenharmonisierungsprozesses und der OHDSI-Tools am Beispiel deutscher GKV-Routinedate in der Praxis untersucht. In diesem Zusammenhang soll eine Evaluation zeigen, ob weitere zusätzliche Prozessschritte zu berücksichtigen sind und inwieweit die abgeleitete Reihenfolge der Prozessschritte in der Praxis umsetzbar ist.
BMC Medical Informatics and Decision Making: https://doi.org/10.1186/s12911-024-02458-7