Mar 13, 2024
Novel protocol advances 3D electron microscopy analysis
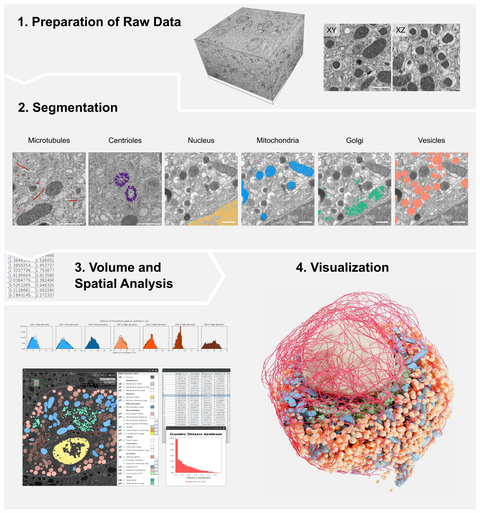
Volume electron microscopy is revolutionizing our knowledge of cells and tissues by enabling structural imaging in 3 dimensions. Now, researchers from the Paul Langerhans Institute Dresden (PLID) of the German Center for Diabetes Research have introduced a novel protocol which could transform the analysis of three-dimensional electron microscopy (3D-EM) datasets. In collaboration with colleagues from the Max Delbrück Center for Molecular Medicine (MDC), the Humboldt-University Berlin, Helmholtz Imaging and the École Polytechnique Fédérale de Lausanne (EFPL) the scientists developed detailed guidelines, aimed at imaging scientists in biology and medicine, that offer a modular and user-friendly workflow to unravel the intricate details of cellular ultrastructure. This new protocol has now been published in the renowned journal “Nature Protocols”.
For decades, two-dimensional electron microscopy has been the cornerstone of cell biology and medical research, providing detailed insights into subcellular components. In recent years, the surge in three-dimensional volume electron microscopy (vEM) methods has led to an abundance of large raw image datasets, necessitating advanced analysis approaches. The newly developed protocol addresses this demand by providing comprehensive procedures for raw data preparation, organelle-specific segmentation, spatial analysis of segmentation maps, and 3D visualization.
Andreas Müller, senior scientist at the PLID and first author of the article elucidates the motivation behind the protocol, stating, “Our aim was to simplify the challenges researchers face when analyzing complete cells in vEM datasets. These datasets are huge and segmentation of the cellular features is very time-consuming. Also, although there are a lot of different software tools available to help with this, not all of them are user-friendly and straight-forward to apply."
The protocol's modular approach stands out as a key feature, offering a versatile framework for vEM analysis. Researchers can choose from a variety of segmentation methods tailored to specific organelles, reducing unnecessary annotation effort, and expediting early project results. Müller emphasizes the adaptability of the workflow, noting, "While the initial project for which we designed the workflow was on pancreatic beta cells, the segmentation, analysis, and rendering workflows are designed to easily adapt to other cell types and vEM modalities."
The advantage of the new protocol is its accessibility to imaging scientists with medium-level computational expertise. The protocol encourages users to incorporate their preferred segmentation tools, providing a streamlined process through “Album” solutions. “Album is a platform that we developed to make complex analysis pipelines easier to distribute and use in a reproducible way.” says Deborah Schmidt, head of the Helmholtz Imaging Solutions Team at MDC and co-first author of the article. This innovative methodology is poised to accelerate advancements in cellular biology, offering a clearer understanding of cellular ultrastructures. Martin Weigert, research group leader at the EFPL and senior author of the protocol summarizes the significance of the protocol, stating, "Our protocol provides an extensive overview of time- and labor-efficient solutions for vEM analysis and should serve as a useful reference for the broader vEM research community."
In summary, this pioneering protocol by Andreas Müller and his co-workers marks a significant stride towards democratizing access to 3D-EM methodologies, fostering collaboration and advancing the frontiers of cellular research.
Original Publication
Müller A, Schmidt D, Albrecht JP, Rieckert L, Otto M, Galicia Garcia LE, Fabig G, Solimena M, Weigert M. Modular segmentation, spatial analysis and visualization of volume electron microscopy datasets. Nat Protoc. 2024 Feb 29. doi: 10.1038/s41596-024-00957-5.