ecgXfusion - Innovative EKG-Analyse mittels vertrauenswürdiger KI
ecgXfusion® – An eXplainable diagnostic support system for reliable ECG analysis. Gewinner des Technology and Innovation Award: Early products and research developments der Europäischen Gesellschaft für Kardiologie (ESC Digital & AI Summit 2025, Berlin). © A. Hammer und M. Schmidt
Unsere Vision
ecgXfusion® - Ein revolutionäres EKG-basiertes Diagnoseunterstützungssystem, das es Ärzten ermöglicht, Herz-Kreislauf-Erkrankungen zuverlässig und nicht-invasiv zu diagnostizieren, indem es vertrauenswürdige und verständliche Diagnosehinweise mit Hilfe von erklärbarer KI liefert.
Zur Umsetzung unserer Vision haben wir am Inkubationsprogramm LifeTechLab teilgenommen, unterstützt durch TUD|excite. Derzeit wird unsere Technologie im Rahmen einer Validierungsförderung bei potenziellen Anwendern im Herzzentrum Dresden und bei niedergelassenen Ärzten in der Praxis getestet und die Key Performance Identifiers (KPIs) quantifiziert.
Unsere Technologien
Vertrauenswürdige KI made in Germany für den Einsatz in der Medizin!
ecgXfusion hat sich zum Ziel gesetzt, KI-Anwendungen für die Analyse von EKG-Signalen zu erschaffen, die sowohl eine hohe Zuverlässigkeit bieten, als auch eine für Ärzte voll transparente Entscheidungsunterstützung liefern und somit nachvollziehbar sind. Damit bieten unsere Technologien das Potenzial nicht nur in der Forschung, sondern auch in der praktizierenden Medizin eingesetzt zu werden.
Unsere Technologien zeichnen sich durch folgende Merkmale aus:
- Selbstständiges Erlernen klinisch relevanter Merkmale
Die KI-Architektur xECGArch kann Morphologie- und Rhythmusmerkmale ohne Vorwissen erlernen und ist dabei hochpräzise und konsistent mit klinischem Lehrbuchwissen. - Intuitive und verständliche Visualisierung
Klinisch relevante Merkmale werden im EKG entsprechend der klinischen Lesart zur Diagnoseunterstützung dargestellt. - Offline Implementierung für personalisierte Anwendungen und Echtzeitlösungen
Die lokale Implementierung von KI-Modellen und Methoden der Individualisierung ermöglichen Anwendungen in der klinischen und ambulanten Versorgung, in der Präzisionsmedizin und in mobilen Anwendungen.
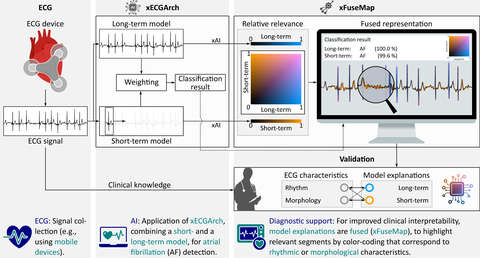
Ansatz zum automatisierten Lernen und Visualisieren klinisch relevanter Langzeit- (rhythmischer) und Kurzzeit- (morphologischer) Merkmale zur Erkennung von Vorhofflimmern (AF) im Elektrokardiogramm (EKG). Es kombiniert die interpretierbare EKG-Architektur (xECGArch) und die beiden darin enthaltenen Modelle (das Langzeit- und das Kurzzeitmodell) mit der fusionierten Darstellung der jeweiligen Modellerklärungen in einer kombinierten Saliency Map (xFuseMap). Die Modellerklärungen wurden auf Übereinstimmung mit EKG-Merkmalen aus dem klinischen Wissen validiert. AI, artificial intelligence; xAI, explainable artificial intelligence. Quelle: A. Hammer u. a., „Fusion of automatically learned rhythm and morphology features matches diagnostic criteria and enhances AI explainability“, npj Artif. Intell. 1, 19 (2025). https://doi.org/10.1038/s44387-025-00022-w.
Die neuartige KI-basierte Modellarchitektur xECGArch® umfasst zwei separate Convolutional Neural Networks (CNN), die sich durch eine unterschiedliche Dimensionierung der Receptive Fields auszeichnen und somit über verschiedene Betrachtungshorizonte verfügen. Dies ermöglicht den Modellen, unterschiedliche Merkmale im Elektrokardiogramm (EKG) zu erlernen und für die Klassifikation zu nutzen. Das Kurzzeit-CNN erlernt schlagmorphologische Merkmale und das Langzeit-CNN Rhythmus. Die Merkmale entsprechen den in der EKG-Analyse typischen Merkmalsgruppen, wodurch eine Interpretation der Merkmale durch Ärztinnen und Ärzte möglich ist.
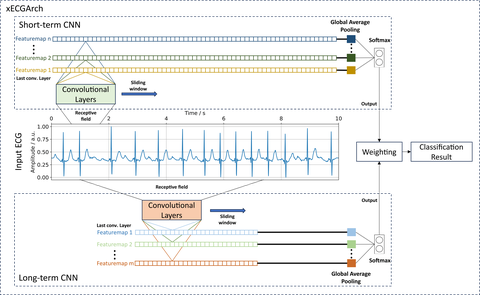
xECGArch® ist eine neuartige Architektur, bestehend aus 2 CNNs, die durch die Dimensionierung der receptive fields unterschiedliche Betrachtungshorizonte haben und unterschiedliche Merkmale (Rhythmus und Morphologie) lernen, siehe https//:xecgarch.com.
Veröffentlichungen:
- M. Goettling, A. Hammer, H. Malberg, und M. Schmidt, „xECGArch: a trustworthy deep learning architecture for interpretable ECG analysis considering short-term and long-term features“, Sci Rep, Bd. 14, Nr. 1, S. 13122, Juni 2024, doi: 10.1038/s41598-024-63656-x.
- A. Hammer, M. Goettling, H. Malberg, A. Linke, und M. Schmidt, „An explainable AI for trustworthy detection of atrial fibrillation on reduced lead ECGs in mobile applications“, European Heart Journal, Bd. 45, Nr. Supplement_1, Okt. 2024, doi: 10.1093/eurheartj/ehae666.3497.
- A. Hammer, H. Malberg, und M. Schmidt, „Morphology Features Self-Learned by Explainable Deep Learning for Atrial Fibrillation Detection Correspond to Fibrillatory Waves“, in 2024 Computing in Cardiology (CinC), S. 1–4., doi: 10.22489/CinC.2024.305.
- A. Hammer, F. Hänsch, L. Seliger, H. Malberg, A. Linke, und M. Schmidt, „Explainable and interpretable AI visualises self-learned clinically relevant ECG characteristics of rhythm and morphology paving the way for trustworthy diagnostic support“, European Heart Journal, Bd. 46, Nr. Supplement_1, Nov. 2025, doi: 10.1093/eurheartj/ehaf784.4412.
- A. Hammer, M. Zannini, H. Malberg, und M. Schmidt, „Transfer Learning to Focus Self -Learning AI on Rhythm Improves Interpretability in Atrial Fibrillation Detection“, in Computing in Cardiology 2025, São Paulo, Brasilien, Sep. 2025, S. 1–4. doi: 10.22489/CinC.2025.396.
xFuseMap® ist eine kombinierte Darstellung der Erklärungen zweier Modelle in einer fusionierten Saliency Map. Saliency Maps sind eine Darstellungsform, bei der die Relevanz von Datenpunkten für die Klassifikationsentscheidung eines Modells durch Einfärbung des Signals angezeigt wird. xFuseMap® ermöglicht die interpretier- und nachvollziehbare Visualisierung von Merkmalen unterschiedlicher Modelle und Merkmalsdomänen. Dazu wird jedem Modell eine eindeutige Farbe zugewiesen. Datenpunkte, die für beide oder keines der Modelle relevant sind, werden in Mischfarben eingefärbt. Dadurch wird ein zweidimensionaler Farbraum mit den Modellen auf jeweils einer Achse aufgespannt.
xFuseMap® erlaubt die kombinierte Darstellung der Erklärungen beider Modelle aus xECGArch® (Lang- und Kurzzeit) sowie der entsprechenden Merkmalsdomänen (Rhythmus und Morphologie) im Elektrokardiogramm (EKG). Morphologie und Rhythmus stellen Standardmerkmale dar, die von Ärztinnen und Ärzten im Rahmen der Diagnostik verwendet und somit eingeordnet werden können. Das Ziel besteht in der Bereitstellung einer möglichst vertrauenswürdigen Diagnoseunterstützung für Ärztinnen und Ärzte.
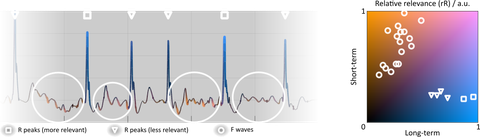
Lesweise der kombinierten Darstellung von Modellerklärungen in einer fusionierten Saliency Map (xFuseMap) anhand eines beispielhaften Elektrokardiogramms (EKG) mit Vorhofflimmern.
Veröffentlichung:
-
A. Hammer, M. Göttling, H. Malberg, A. Linke, S. Richter, N. Mangner und M. Schmidt, „Fusion of automatically learned rhythm and morphology features matches diagnostic criteria and enhances AI explainability“, npj Artificial Intelligence 1, 19 (2025). https://doi.org/10.1038/s44387-025-00022-w.
- A. Hammer, F. Hänsch, L. Seliger, H. Malberg, A. Linke, und M. Schmidt, „Explainable and interpretable AI visualises self-learned clinically relevant ECG characteristics of rhythm and morphology paving the way for trustworthy diagnostic support“, European Heart Journal, Bd. 46, Nr. Supplement_1, Nov. 2025, doi: 10.1093/eurheartj/ehaf784.4412.
Das autonome Nervensystem (ANS) kontrolliert zahlreiche Körperfunktionen und zeigt je nach persönlichem Zustand spezifische Reaktionen. Die Bewertung des ANS erlaubt daher weitreichende Aussagen, beispielsweise zum Risiko lebensbedrohliche Erkrankungen zu erleiden, eine Bewertung des Schlafs und die Erfassung der Stressbelastung.
Das Verfahren Two-Dimensional Signal Warping (2DSW) bietet die Möglichkeit komplexe morphologische Veränderungen zwischen Herzschlägen zu erfassen und derartige Aussagen nicht-invasiv treffen zu können (z.B. durch die Analyse der QT-Variabilität). Ziel ist eine wesentliche Verbesserung der diagnostischen Möglichen, insbesondere auch die Prädiktion und Prävention, ohne dass für die Patienten eine zusätzliche Belastung entsteht.
Weitere Informationen sind auf 2dsw.com zu finden.
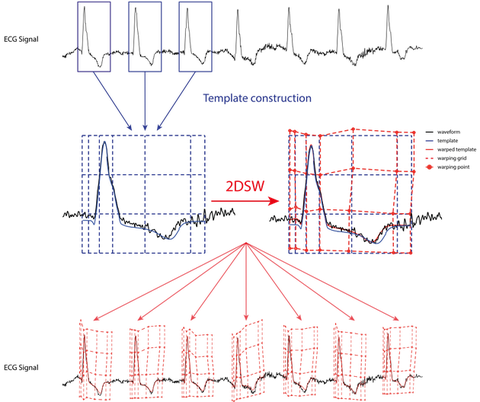
Schema des Two-Dimensional Signal Warping (2DSW, siehe auch http://2dsw.com/).
Veröffentlichungen:
- M. Schmidt, M. Baumert, A. Porta, H. Malberg, und S. Zaunseder, „Two-Dimensional Warping for One-Dimensional Signals—Conceptual Framework and Application to ECG Processing“, IEEE Transactions on Signal Processing, Bd. 62, Nr. 21, S. 5577–5588, Nov. 2014, doi: 10.1109/TSP.2014.2354313.
-
M. Schmidt, M. Baumert, H. Malberg, und S. Zaunseder, „Iterative two-dimensional signal warping—Towards a generalized approach for adaption of one-dimensional signals“, Biomedical Signal Processing and Control, Bd. 43, S. 311–319, Mai 2018, doi: 10.1016/j.bspc.2018.03.016.
-
Weitere Publikationen unter 2dsw.com
Klinische Partner
Unser IP
Das Patent DE 10 2023 118 246.3/PCT DE2024 100599/EP 24 745 624.7/US 19/499,953 umfasst das Prinzip von xFuseMap und die KI-Architektur von xECGArch. Patentanmeldung EP 25 175 552.6 umfasst Methoden zur Individualisierung der KI-Modelle. Das erteilte Patent DE 10 2014 100 609.7 umfasste die Architektur von 2DSW.
ecgXfusion ist eine Initiative der AG Biosignalverarbeitung des Insituts für Biomedizinische Technik und kann so auf eine langjährige wissenschaftliche Expertise im Bereich Erfassung, Verarbeitung und Analyse von Biosignalen mit unterschiedlichsten Methoden und Technologien zurückgreifen.
Team

Doktorand | ecgXfusion
NameHerr Dipl.-Wi.-Ing. Alexander Hammer
AG Biosignalverarbeitung
Eine verschlüsselte E-Mail über das SecureMail-Portal versenden (nur für TUD-externe Personen).
Besuchsadresse:
Fetscherforum (29), 1. OG, Raum 30 Fetscherstraße 29
01307 Dresden

Arbeitsgruppenleiter
NameHerr Dr.-Ing. Martin Schmidt
AG Biosignalverarbeitung
Eine verschlüsselte E-Mail über das SecureMail-Portal versenden (nur für TUD-externe Personen).
Besuchsadresse:
Fetscherforum (F29), 1.OG, Raum 34 Fetscherstraße 29
01307 Dresden
Patronage
 © H. Malberg
© H. Malberg
Institutsdirektor
NameHerr Prof. Dr.-Ing. habil. Hagen Malberg
Eine verschlüsselte E-Mail über das SecureMail-Portal versenden (nur für TUD-externe Personen).
Besucheradresse:
Fetscherforum (F29), 1. OG, Raum 11 Fetscherstr. 29
01307 Dresden
None
Ärztlicher Direktor des Herzzentrums Dresden
NameHerr Prof. Dr. med. habil. Axel Linke





















