Forschung
Forschung im Bringmann-Labor: Steuerung und Funktionen des Schlafs
Schlaf ist ein systemischer physiologischer Zustand, der für das Überleben eines Organismus unerlässlich ist. Schlaf beeinflusst praktisch alle physiologischen Prozesse und spielt eine herausragende Rolle bei Entwicklung, Gewebehomöostase, Regenerationsprozessen und Langlebigkeit [1, 2]. Schlaflosigkeit und Schlafstörungen sind in modernen Gesellschaften weit verbreitet und stellen ein massives ungelöstes Gesundheitsproblem dar [3]. Alle Tiere mit einem Nervensystem schlafen, was darauf hindeutet, dass dieser physiologische Zustand früh in der Evolution entstanden ist [4]. Um den Schlaf zu verstehen, müssen wir verstehen, wie er gesteuert wird und wie er seine Funktionen auf molekularer und zellulärer Ebene ausübt. Um diese Fragen zu lösen, hat sich unser Labor auf eine langfristige Forschungsreise begeben, um zunächst herauszufinden, wie der Schlaf bei C. elegans gesteuert wird. Das molekulare Wissen über den Schlaf nutzen wir dann, um C. elegans genetisch den Schlaf zu entziehen und so die molekularen Mechanismen zu verstehen, die seinen zellbiologischen und biochemischen Funktionen zugrunde liegen. Schließlich nutzen wir die molekularen Schlafinformationen von C. elegans als Fahrplan, um auch den Schlaf von Säugetieren zu verstehen. Zusammenfassend haben wir drei große Forschungsziele, die wir verfolgen:
Ziel 1. Wir möchten ein molekulares Verständnis der Mechanismen der Schlafregulation bei C. elegans erlangen
Ziel 2. Wir möchten die lebenswichtigen Funktionen des Schlafs auf molekularer Ebene bei C. elegans verstehen
Ziel 3. Wir möchten das Schlafwissen von C. elegans nutzen, um den Schlaf von Säugetieren zu verstehen
ZIEL 1. Wir möchten ein molekulares Verständnis der Mechanismen der Schlafregulation bei C. elegans erlangen.
Es ist wenig darüber bekannt, wie der Schlaf auf molekularer Ebene gesteuert wird. Es ist wichtig, die Kontrollmechanismen des Schlafs zu verstehen, da dies Ansatzpunkte für die Manipulation des Schlafs bietet, um seine Funktionen zu untersuchen und Behandlungen für Schlafstörungen zu entwickeln. Molekulare Informationen zur Schlafregulation werden die Übertragung der Erkenntnisse auf andere Organismen und die Entwicklung von Therapien für menschliche Schlafstörungen erleichtern. Der Schlaf bei Säugetieren wird durch GABAerge Peptiderge Neuronen induziert, die zu Beginn des Schlafes depolarisieren und daher als „schlafaktive“ Neuronen bezeichnet werden [5]. Die molekulare Kontrolle schlafaktiver Neuronen ist jedoch nicht gut verstanden. Um zu verstehen wie der Schlaf gesteuert wird, müssen wir verstehen, wie die Depolarisation schlafaktiver Neuronen reguliert wird. Dazu gehören die biochemischen Signalwege, die das Schlafbedürfnis nach längerem Wachzustand und einen erhöhten Schlafbedarf während der Entwicklung, Regeneration oder Krankheit signalisieren. Wir haben uns zum Ziel gesetzt, C. elegans als Modell zur Untersuchung molekularbiologischer Mechanismen schlafaktiver Neurone zu entwickeln. Da Schlaf lebenswichtig ist, kommt er in jedem Tier vor, welches ein Nervensystem besitzt, von der Qualle bis zum Menschen [6]. Schlaf kann anhand von Verhaltenskriterien wie verminderte Reaktionsfähigkeit auf Stimulation, Reversibilität und homöostatische Regulation identifiziert werden [7]. C. elegans zeigt Schlafverhalten in allen Lebensstadien und unter verschiedenen Bedingungen. Unser Labor war das erste, das ein systematisches genetisches Screening auf Schlafgene in C. elegans durchgeführt hat [8]. Wir fanden eine Reihe von Genen, die für den Schlaf erforderlich sind, und die Analyse dieser Gene führte zur Identifizierung eines einzelnen schlafaktiven Neurons in C. elegans namens RIS. Das RIS-Neuron verhält sich wie schlafaktive Neuronen von Säugetieren: Es depolarisiert zu Beginn des Schlafs und induziert aktiv den Schlaf, indem es Wachschaltkreise durch die Freisetzung von GABA und Neuropeptiden hemmt (Abbildung 1-2) [9].
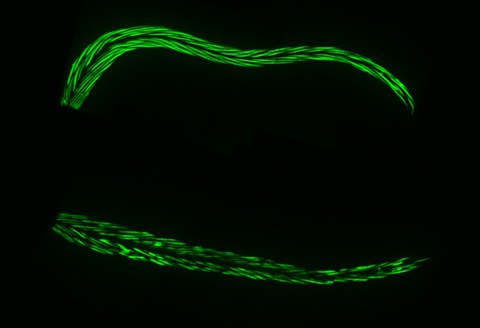
Abbildung 1. Das RIS-Neuron ist während des Schlafs aktiv. Hier wurde die Aktivität aller Neuronen mit einem genetisch kodierten Kalziumindikator namens GCaMP visualisiert (grüne Fluoreszenz, je heller das grüne Licht, desto aktiver das Neuron). Oben: Im Wachzustand sind die meisten Neuronen die meiste Zeit aktiv. Unten: Während des Schlafs sind die meisten Neuronen inaktiv, mit Ausnahme des RIS-Neurons, das spezifisch im Schlaf aktiviert wird.
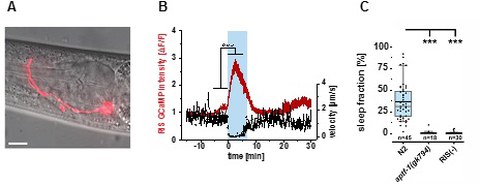
Abbildung 2. Das schlafaktive RIS-Neuron induziert den Schlaf. A) Das einzelne schlafaktive RIS-Neuron (rot). B) RIS depolarisiert während Schlafphasen. C) RIS ist für den Schlaf notwendig.
Die Identifizierung eines einzelnen schlafaktiven Neurons in C. elegans bietet eine einzigartige Gelegenheit, die Molekularbiologie der Schlafkontrolle in einem zugänglichen Modellsystem zu untersuchen. Die aktuellen Arbeiten in unserem Labor konzentrieren sich auf Signalwege, die RIS aktivieren, darunter antimikrobielle Peptide, die als Reaktion auf Verletzungen oder Infektionen produziert werden [10], EGF-Signalwege, die während der Entwicklung und bei Stress aktiviert werden [11], sowie konservierte, die Langlebigkeit fördernde Signalwege wie FOXO/DAF-16 und AMP-aktivierte Kinase [12].
ZIEL 2. Wir möchten die lebenswichtigen Funktionen des Schlafs auf molekularer Ebene bei C. elegans verstehen
Das Wissen darüber, wie der Schlaf gesteuert wird, ermöglicht die Manipulation dieses Zustands und damit das Studium der Folgen von zu viel oder zu wenig Schlaf. Da der Schlaf in C. elegans zwingend das einzelne RIS-Neuron benötigt, kann der Schlaf durch Ablation von RIS einfach und effizient aus diesem System entfernt werden. Umgekehrt kann die Erhöhung der Aktivität dieses Neurons, beispielsweise durch Optogenetik, zur Steigerung des Schlafs genutzt werden. Somit kann der Schlaf durch Manipulation schlafaktiver Neuronen kontrolliert werden, was die Untersuchung der Phänotypen ermöglicht, die entweder durch Schlafverlust oder erhöhten Schlaf verursacht werden.
Unser schlafloses C. elegans-System bietet eine einzigartige Chance, die Folgen von Schlafverlust und damit die molekularen Schlaffunktionen an einem zugänglichen Modelltier zu untersuchen. Unser Labor analysiert schlaflose Würmer und wir haben herausgefunden, dass Schlaf den Alterungsprozesses verlangsamt [12],und hilft, Verletzungen zu überleben [10]. Ein hochkonserviertes Netzwerk von Genen steuert das Altern [7]. Wir zeigten, dass dieses alternde Gennetzwerk die Aktivität des schlafaktiven RIS-Neurons durch FoxO und AMP-aktivierte Kinase steuert, um den Schlaf zu induzieren. Die Depolarisation von RIS wiederum induziert Schlaf, um das Fortschreiten der alternden Phänotypen zu verlangsamen. Anstatt Energie für die spätere Verwendung zu sparen, ist Schlaf also Teil eines Schutzprogramms, das von Anti-Aging-Genen induziert wird, um der Zellalterung entgegenzuwirken (Abbildung 3-4) [12].
Abbildung 3. RIS hemmt Alterungsprozesse. Eine Zusammenfassung des Zusammenhangs zwischen Altern, Hunger und Schlaf. Nährstoffmangel hemmt Wachstumsprozesse durch ein konserviertes Langlebigkeitsgennetzwerk, welches auch RIS depolarisiert. RIS wiederum wirkt dem Fortschreiten des Alterungsprozesses entgegen und verhindert so den Tod. Wie RIS wirkt, um den Alterungsprozess zu verlangsamen, ist Gegenstand aktueller Studien.
Abbildung 4. Beeinträchtigung des schlafaktiven RIS-Neurons vermindert die Gesundheit und beschleunigt den Alterungsprozess. Der Wurm oben exprimiert einen Muskelmyosin-Reporter und schläft regelmäßig. Es ist gesund und seine Muskelfasern sind gut erhalten. Dem Wurm unten im Bild fehlt es an Schlaf, weil er eine Mutation trägt, die das schlafaktive RIS-Neuron beeinträchtigt. Als Folge der RIS-Beeinträchtigung werden Muskelfasern schneller zerstört. Ein solcher Muskelabbau wird typischerweise während des Alterns bei allen Arten beobachtet, einschließlich beim Menschen, wo dieser Zustand als Sarkopenie bezeichnet wird.
ZIEL 3. Wir möchten das Schlafwissen von C. elegans nutzen, um den Schlaf von Säugetieren zu verstehen
Schlaf ist bei Tieren weit verbreitet und wurde bei allen Tieren gefunden, die ein Nervensystem haben. Molekulargenetik und neuronale Schaltkreisanalysen haben gezeigt, dass die der Schlafregulation zugrunde liegenden Mechanismen bei allen Arten hoch konserviert sind. Zum Beispiel wird Schlaf in allen Tieren durch schlafaktive schlaffördernde Neuronen induziert, die durch GABA und Neuropeptide direkt die neuronalen Wachseins-Schaltkreise hemmen [4]. Auch Gene, die den Schlaf steuern, sind konserviert, ein Beispiel sind AP2-Transkriptionsfaktoren [8, 13-15]. Unser Labor untersucht den Schlaf von Mäusen in Mutanten von Genen, die wir in Würmern identifiziert haben. Wir haben diese Forschungslinie mit der Untersuchung der Rolle von AP2-Transkriptionsfaktoren im Mausschlaf begonnen (Abbildung 5). Bei Säugetieren diversifizierte sich AP2 in mehrere TFAP2-Proteine, die von getrennten Genen (TFAB2A, TFAB2b usw.) kodiert werden. Unsere Ergebnisse von Schlaf-Messungen bei AP2-mutierten Mäusen zeigten, dass AP-2-Transkriptionsfaktoren auch bei Mäusen das Schlafverhalten steuern, dass aber die Rolle der AP-2-Gene funktionell diversifiziert ist, um eine bidirektionale Kontrolle der Schlafqualität zu ermöglichen. Dabei fördert TFAP2B den Schlaf und TFAP2A hemmt den Schlaf. Diese Divergenz der Funktionen des AP-2-Transkriptionsfaktors könnte möglicherweise die Evolution komplexerer Schlaftypen unterstützt haben [16].
Abbildung 5: Eine C57BL/6-Maus schläft in unserem Labor.
Wir untersuchen Gene, die den Schlaf in C. elegans kontrollieren, auch in Mäusen.
Literaturverweise
1. Cirelli, C., and Tononi, G. (2008). Is sleep essential? PLoS biology 6, e216.
2. Mignot, E. (2008). Why we sleep: the temporal organization of recovery. PLoS biology 6, e106.
4. Bringmann, H. (2018). Sleep-Active Neurons: Conserved Motors of Sleep. Genetics 208, 1279-1289.