13.04.2017
Dresdner Forscher entwickeln Algorithmus zur automatischen Erkennung signifikanter Zusammenhänge latenter Variablen im Bereich Big Data

Forschungsgruppe von Dr. Carlo Vittorio Cannistraci
Die von Dr. Carlo Vittorio Cannistraci, Leiter der Forschungsgruppe ‘Biomedical Cybernetics’ am BIOTEChnologischen Zentrum TU Dresden, geleitete internationale Forschergruppe hat ‚PC-corr‘ entwickelt: einen intelligenten Algorithmus, der Kerngruppen interagierender latenter Variablen (welche Unterschiede in Big Data erzeugen) automatisch entdecken kann. PC-corr fand wichtige molekulare Signaturen in mehr als sechs verschiedenen Feldern im Bereich der OMIC-Wissenschaften (wie Lipidomik, Metagenomik, Genomik, Mechanomik). Dies stellt einen entscheidenden Schritt in Richtung der Entdeckung kombinatorischer Biomarker in der Präzisionsmedizin dar.
Algorithmen sind eigenständige Handlungsvorschriften zur Lösung eines bestimmten Problems. Im Hinblick auf die gegenwärtige Computing- Revolution, gibt es eine bestimmte Klasse von Algorithmen die ‘Intelligente Algorithmen’ genannt werden. Diese sind in der Lage, die für ein intelligentes Verhalten notwendigen fortgeschrittenen menschliche Fähigkeiten nachzubilden. Ein gut ausgebildeter und erfahrener Arzt ist z.B. in der Lage, Zusammenhänge zwischen einem Set an klinischen Variablen zu erkennen um daraus eine Beschreibung und Diagnose der krankhaften Zustände im Körper eines Patienten ableiten zu können. Die Diagnose wird hierbei vor allem durch das Verbinden von Kombinationen unterschiedlicher Variablen gestärkt. Diese Art intelligenter Logik wird von ‚PC-corr‘ nachgebildet – einem Algoritmus der von Dr. Cannistraci erdacht und von der Biomedical Cybernetics Forschungsgruppe am BIOTEC realisiert und getestet wurde. Das Team bestand dabei aus internationalen Wissenschaftlern aus mehr als sechs verschiedenen Feldern der Omic-Wissenschaft (wie z.B. Lipidomik, Metagenomik, Genomic und Mechanomik). Die Studie wurde von der Klaus Tschira Stiftung gGmbH (KTS) unterstützt und umfasst Kooperationen mehrerer akademischer Partner, wie z.B. der Forschungsgruppe Cellular Machines am BIOTEC, dem Center for Regenerative Therapies TU Dresden (CRTD), der Forschungsgruppe von Andrej Shevchenko am Max Planck Institute of Molecular Cell Biology and Genetics (MPI-CBG), sowie dem Department of Stem Cell Biology an der University of Nottingham (UK) und der Integrin Signalling Gruppe am Fundación Centro Nacional de Investigaciones Cardiovasculares Carlos III (CNIC) in Madrid (Spanien). Entscheidend war auch die Mitwirkung der Lipotype GmbH, einem Industriepartner und Experten im Bereich der Lipidomik für Gesundheit. Die Studie konnte außerdem auf Datensätze des RIKEN Omics Science Center (OSC) in Yokohama (Japan) und des FANTOM consortium zurückgreifen.
Wenn PC-corr auf einen sehr großen Datensatz mit vielen Variablen (z.B. Big Data) angewendet wird, dann nutzt der Algorithmus eine unbeaufsichtigte Maschinenlernen-Analyse, um automatisch Kohorten aus Proben zu identifizieren, die verschiedenen Trends im multidimensionalen Variablenraum zugrunde liegen. Auderdem lokalisiert PC-corr, wie die Variablen (z.B. Ebenen verschiedener Lipide im Blutplasma) sich zu Subnetz-Modulen (welche der Proben/Patienten-Diskriminierung zugrunde liegen) zusammenschließen und sich selbst organisieren. Anders als die bisher für die Biomedizin entwickelten Algorithmen (die nur auf genomische Analysen fokussiert sind), kann der intelligente Algoroithmus PC-corr bei jeder Art von Datensatz angewendet werden. Dies schließt vielfältige, große molekulare Datensätze ein und zudem können Faktor-Kombinationen zur Erklärung biologischer Unterschiede zwischen Proben/Patienten hervorgehoben werden. Der Algorithmus wurde an Big Data aus verschiedenen Bereichen der Omic-Wissenschaft getestet, was auch Felder der Molekularbiologie beinhaltet – die eine Charakterisierung eines biologischen Systems als ein großes Ensemble homogener molekularer Eigenschaften generiert. So widmet sich z.B. der Bereich der Lipidomik der Untersuchung zellulärer Lipide in einem biologischen System, während sich der Bereich der Proteomik der Untersuchung aller Proteine in einem biologischen System widmet.
Der Input für PC-corr ist ein großer Datensatz, der eine große Anzahl an Variablen enthält. Ausgegeben wird ein visuelles Netzwerk, welches die signifikanten Verbindungen illustriert. Zugrunde liegen hierbei die biolgischen Unterschiede zwischen den Proben. Wenn zum Beispiel ein großer genomischer, biomedizinischer Datensatz analysiert wird, gibt PC-corr eine reduzierte genetische Signatur aus tausenden von Genen aus. Diese kann dann genutzt werden, um kombinatorische und Multiskalen-Biomarker zu gestalten (siehe Bild 2). Biomarker im Bereich der Biomedizin beziehen sich auf jede messbare biologische Charakteristik, wie z.B. Moleküle oder klinische Variablen – welche dann als Indikatoren für einen biologischen Status, Zustand oder Prozess genutzt werden können. Sie können sowohl in der Grundlagenforschung als auch in der klinischen Forschung genutzt werden, um prognostische Informationen bereitzustellen oder die Wirkung von Medikamenten während einer Behandlung zu untersuchen.
“In einem der analysierten Datensätze konnten die durch PC-corr entdeckten Teilnetzwerke genetischer Variationen große genetische Unterschiede zwischen zwei japanischen Bevölkerungsgruppen (eine aus Tokio und eine aus Okinawa) erklären und damit neuartige kombinatorische Assoziationen zwischen Erkrankungsrisiko-bezogenen Varianten bei Tokiotern bereitstellen“, erklärt Sara Ciucci, Erstautorin der Studie.
“Unser Algorithmus hat die einzigartige Besonderheit, dass er Variablen des selben biologischen Systems kombinieren kann – selbst wenn diese mit unterschiedlichen Skalen gemessen wurden. In unserer Mechanomik-Studie haben wir zum Beispiel genomische und mechanische Variablen der Krebs-Stammzellen von Patienten mit einander kombiniert. In den nächsten Monaten werden wir den Algorithmus weiterentwickeln. Unser langfristiges Ziel ist die Entwicklung eines Artificial Intelligence (AI) ‘wissenschaftlichen Assistenten‘ – der direkt mit Forschern interagieren, Daten automatisch analysieren (ohne menschliche Programmierung) und ein automatisches, kombinatorisches und Multiskalen-Design bereitstellen kann“, so Carlo Vittorio Cannistraci, der Korrespondenz-Autor der Studie.
Carlo Vittorio Cannistraci ist ein Theoretischer Ingenieur. Seit Februar 2014 leitet er die Forschungsgruppe ‚Biomedical Cybernetics‘ am BIOTEC und seit 2016 ist er TUD Young Investigator in der Fachrichtung Physik an der TU Dresden. Zu seinen Forschungsinteressen zählen Themen im Grenzbereich zwischen Physik und komplexer Systeme, komplexer Netzwerke und Maschinenlernen-Theorie. Zuvor (von 2010-2013) arbeitete er als Forscher und Postdoktorand an der King Abdullah University of Science and Technology (KAUST, Jeddah, Saudi Arabien) und der University of California at San Diego (UCSD). Seinen Doktortitel erwarb Carlo Cannistraci von der Scuola Interpoltecnica di Dottorato (SIPD, Mailand, Italien) – wo er im Bereich der Biomedizintechnik arbeitete und sich dabei auf komplexe Netzwerke, intelligente Systeme und Maschinenlernen in der Biomedizin konzentrierte (2007-2009).
Sara Ciucci promoviert derzeit in der Forschungsgruppe von Dr. Carlo Cannistraci. Der Fokus ihrer Arbeit liegt auf dem maschinellen Lernen und auf Techniken der Netzwerk Biologie zur Erforschung und Analyse von OMIC-Daten. Ihr Masterstudium hat sie an der University of Trento in Mathematics absolviert (Abschluss 2014).
Publikation
“Enlightening discriminative network functional modules behind Principal Component Analysis separation in differential-omic science studies”
Sara Ciucci, Yan Ge, Claudio Durán, Alessandra Palladini, Víctor Jiménez-Jiménez, Luisa María Martínez-Sánchez, Yuting Wang, Susanne Sales, Andrej Shevchenko, Steven W. Poser, Maik Herbig, Oliver Otto, Andreas Androutsellis-Theotokis, Jochen Guck, Mathias J. Gerl & Carlo Vittorio Cannistraci
Scientific Reports 7, Artikel-Nummer: 43946 (2017)
DOI: 10.1038/srep43946
http://www.nature.com/articles/srep43946
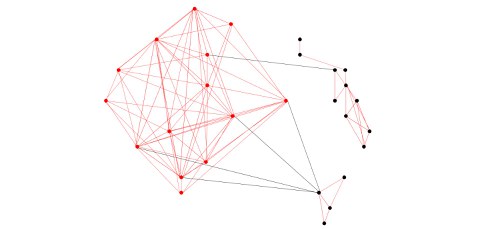
Exemplarische Darstellung kombinatorischer und skalenübergreifender Biomarker
Informationen für Journalisten:
Franziska Clauß, M.A.
Pressesprecherin
Tel.: +49 (0) 351 458 82065
Das Biotechnologische Zentrum (BIOTEC) wurde 2000 als zentrale wissenschaftliche Einrichtung der Technischen Universität Dresden mit dem Ziel gegründet, modernste Forschungsansätze in der Molekular- und Zellbiologie mit den in Dresden traditionell starken Ingenieurswissenschaften zu verbinden. Innerhalb der TU Dresden nimmt das BIOTEC eine zentrale Position in Forschung und Lehre mit dem Schwerpunkt „Molecular Bioengineering und Regenerative Medizin“ ein. Es trägt damit entscheidend zur Profilierung der TU Dresden im Bereich moderner Biotechnologie und Biomedizin bei. Die Forschungsschwerpunkte der internationalen Arbeitsgruppen bilden die Zellbiologie, Nanobiotechnologie und die Bioinformatik. https://www.biotec.tu-dresden.de
