BMBF e:Med HaematoOPT "Modell-basierte Optimierung und Individualisierung von Behandlungsstrategien in der Hämatologie"
Die modellbasierte Optimierung und Individualisierung der Behandlungsstrategien in der Hämatologie
Im interdisziplinären Konsortium HaematoOPT haben sich Mathematiker, Statistiker und Bioinformatiker mit Partner aus führenden klinischen Studiengruppen, ärzten und Biologen zusammengeschlossen. Das Ziel dieser Kooperation ist es, an ausgewählten Beispielen zu demonstrieren, dass es sinnvoll und möglich ist, existierende mathematische Modelle physiologischer und pathophysiologischer Prozesse im Rahmen klinischer Entscheidungsprozesse nutzbar zu machen und somit zu einer Optimierung von Therapiestrategien durch die Anwendung system-medizinischer Techniken beizutragen.
Das HaematoOPT-Konsortium ist Teil der e:Med Initiative des Bundesministeriums für Bildung und Forschung (BMBF).
Koordiniert wird dieses Verbundprojekt von Prof. Dr. Ingo Röder am Institut für Medizinische Informatik und Biometrie an der TU Dresden (TUD). Weitere am Projekt beteiligte Einrichtungen sind das Universitätsklinikum Carl Gustav Carus Dresden, MK I (UKD), die Universität Leipzig, IMISE (UL), das Universitätsklinikum Jena (UKJ), das Universitätsklinikum Köln (UKK), sowie das Fritz-Lipmann-Institut in Jena (FLI).
Das BMBF-geförderte Projekt HaematoOPT zielt darauf ab, zu zeigen, dass Modelle von normaler und leukämischer Hämatopoese verwendet werden können, um die klinische Entscheidungsfindung geeignet zu beeinflussen. Das Projekt baut auf den Modellen der großen drei hämatopoetischen Zelllinien auf (z.B. Erythropoese, Granulozytopoese, Thrompopoiese und ihren regulatorischen Wachstumsfaktoren), um die klonale Zellexpansion für hämatopoetische Stammzellen, sowie den darauf aufbauenden Ausbruch und die Behandlung klonaler Krankheiten wie z.B. die chronisch-myeloische Leukämie (CML), zu beschreiben. Unser interdisziplinäres Konsortium vereint mathematische Modellierer und Bioinformatiker mit klinischen Hämatologen und Biologen. Es ist unser übergeordnetes Ziel, das klinische Potential der zuvor entwickelten mathematischen Modelle für die Behandlungsoptimierung und -individualisierung im Bereich der Hämatologie zu demonstrieren.
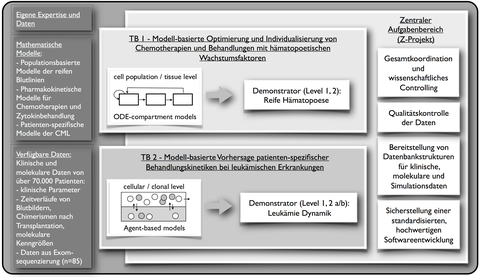
Zentrales Ziel dieses Projektes ist die Demonstration der praktischen Anwendbarkeit mathematischer Modelle zur Beschreibung normaler und leukämischer Blutbildung im Rahmen klinischer Entscheidungsprozesse. Das Projekt basiert maßgeblich auf Modellen, die innerhalb der letzten Jahre in beteiligten Arbeitsgruppen entwickelten worden. Hierbei handelt es sich um mathematische Modelle der drei Hauptentwicklungslinien hämatopoetischer Zellen (d.h. der Erythropoese, Granulopoese, Thrombopoese) und deren Regulation durch Wachstumsfaktoren sowie um Modelle zur Beschreibung hämatopoetischer Stammzellen. Letztere wurden bereits zur Beschreibung der Entwicklung und des Behandlungsverlaufes klonaler Erkrankungen des blutbildenden Systems (z.B. chronische myeloische Leukämie, CML) eingesetzt.
Diese Modelle, die in enger Kooperation zwischen Grundlagenforschern und klinischen Studiengruppen in der letzten zehn Jahren entwickelt und konsolidiert wurden, sind nunmehr auf einem Entwicklungsstand, welcher es realistisch erscheinen lässt, sie auch im Rahmen klinischer Entscheidungsprozesse praktisch anzuwenden. Um dies zu ermöglichen, ist sowohl eine Weiterentwicklung wie auch eine Anpassung entsprechend neuer biologischer Erkenntnisse notwendig. Speziell werden wir die Modelle zukünftig dahingehend erweitern, dass eine explizite Beschreibung der Patienten- und Therapieheterogenität inklusive individueller Risikofaktoren möglich ist, um Risiken adaptierter Therapien besser abschätzen und individualisierte Therapiekonzepte entwickeln zu können.
Das vorgeschlagene, interdisziplinäre Konsortium vereint Mathematiker und Bioinformatiker mit aktiv klinisch arbeitenden Hämatologen sowie Biologen. Hierbei bauen wir auf einer Reihe existierender, langjähriger Kooperationen zwischen einigen Mitgliedern des Konsortiums auf. Weiterhin konnte bereits in der Vergangenheit gezeigt werden, dass modellbasierte Studienplanung im Bereich der Behandlung von Lymphomen und Leukämien nutzbringend zur Erzielung verbesserter Behandlungsergebnisse eingesetzt werden kann. Einige Partner sind federführend in internationalen Studiengruppen involviert; ein Fakt der es unserem Konsortium ermöglicht, Zugriff auf neueste Studiendaten zu erhalten bzw. auf direktem Wege zur Planung und Initiierung neuer Studien beizutragen.
Aufbauend auf diesen sehr guten strukturellen Voraussetzungen ist geplant, die vorhandenen Modelle so zu erweitern und aufzuarbeiten, dass ihr klinischer Nutzen sowie ihre praktische Anwendbarkeit konkret demonstriert werden kann. Weiterhin streben wir an, die Modellierung und Modellvalidierung in einer Art und Weise zu realisieren, die es ermöglicht, zu einem späteren Zeitpunkt zertifizierte Softwareprodukte zu entwickeln.
Koordination: Prof. Dr. Ingo Röder, Institut für medizinische Informatik und Biometrie, Technische Universität Dresden
Stellv. Koordination: Prof. Dr. Markus Löffler, Institut für medizinische Informatik, Statistik und Epidemiologie, Universität Leipzig
Dabei werden wir uns auf zwei Hauptinteressengebiete konzentrieren:
- Optimierung der Wachstumsfaktor-Behandlung gestörter normale Hämatopoiese und
- Optimierung der Leukämie-Behandlung bei akuter und chronisch-myeloischer Leukämie (CML).
Themenbereiche
-
Themenbereich 1: Modell-basierte Optimierung und Individualisierung von Chemotherapien und Behandlungen mit hämatopoetischen Wachstumsfaktoren
-
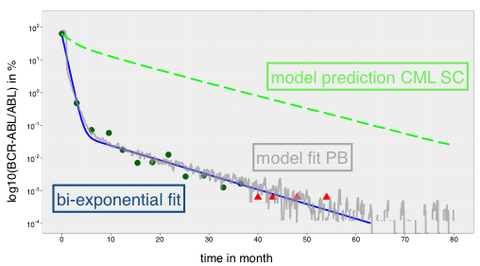
Themenbereich 2: Modell-basierte Vorhersage patienten-spezifischer Behandlungskinetiken bei leukämischen Erkrankungen
-
Themenbereich Z: Das Zentralprojekt (Z) stellt eine konsortiumübergreifende Plattform dar, welche die Partner des HaematoOPT-Projektes unterstützt
Beteiligte Wissenschaftler
- Christoph Baldow
-
Hendrik Liebscher
-
Sebastian Wagner
Publikationen
Finanzierung
BMBF (031A424A) Förderung innerhalb der e:Med Initiative -- Modul II - Demonstratoren für die individualisierte Medizin